论文作者:施国邦,指导教师:郑珩
所属学院:生命科学与技术学院,二级学科:微生物与生化药学
01 导师说
 施国邦同学的硕士毕业论文获得省级优秀论文,和他的努力以及创新思维是分不开的。当前多肽序列记录格式无法直接体现序列上的修饰信息,难点在于当前的序列比对软件无法辨认20种天然氨基酸的单字母缩写外的其它字符。该同学创造性地引入了封闭式字母标记法,并保留对应母肽序列用于搜索比较,使得订书肽修饰位点可直观显示,且不影响序列的检索和分析,对于修饰多肽的记录提供了新的模式。论文不仅更新了DRAMP数据库,还进行了多肽结构预测,构建了抗菌肽活性预测模型等,促进了新型抗菌肽的设计,对机器学习与人工智能在药物设计领域应用作出了积极探索。这项研究也体现了跨学科交叉的重要性,多个领域的融合使得这项研究得以顺利推进并取得显著成效,希望同学们能注重跨学科知识的学习,获得更多的创新成果。
施国邦同学的硕士毕业论文获得省级优秀论文,和他的努力以及创新思维是分不开的。当前多肽序列记录格式无法直接体现序列上的修饰信息,难点在于当前的序列比对软件无法辨认20种天然氨基酸的单字母缩写外的其它字符。该同学创造性地引入了封闭式字母标记法,并保留对应母肽序列用于搜索比较,使得订书肽修饰位点可直观显示,且不影响序列的检索和分析,对于修饰多肽的记录提供了新的模式。论文不仅更新了DRAMP数据库,还进行了多肽结构预测,构建了抗菌肽活性预测模型等,促进了新型抗菌肽的设计,对机器学习与人工智能在药物设计领域应用作出了积极探索。这项研究也体现了跨学科交叉的重要性,多个领域的融合使得这项研究得以顺利推进并取得显著成效,希望同学们能注重跨学科知识的学习,获得更多的创新成果。
02 作者说
 评审对我们课题组工作成果的肯定是我能获奖的直接原因,根本原因是我们课题组多位成员近十年的积累与沉淀。课题组从零开始收集数据、搭建抗菌肽数据库及网站,到后来一步步完善数据库,并利用丰富的数据来指导抗菌肽的设计,之后课题组的成员又进行湿实验的验证,一切都是连贯有逻辑的。数据的收集和利用横亘了我的整篇论文,而有可能是之后生物学领域发展的某条脉络,人工智能在其中成为了重要的抓手工具。不管是药物研究与发现、精准医疗、基因组学研究、细胞生物学,以丰富的海量数据为基础,佐以实验的验证,有大量可供挖掘探索的科研课题,请师弟师妹们抓住机会大展宏图吧!
评审对我们课题组工作成果的肯定是我能获奖的直接原因,根本原因是我们课题组多位成员近十年的积累与沉淀。课题组从零开始收集数据、搭建抗菌肽数据库及网站,到后来一步步完善数据库,并利用丰富的数据来指导抗菌肽的设计,之后课题组的成员又进行湿实验的验证,一切都是连贯有逻辑的。数据的收集和利用横亘了我的整篇论文,而有可能是之后生物学领域发展的某条脉络,人工智能在其中成为了重要的抓手工具。不管是药物研究与发现、精准医疗、基因组学研究、细胞生物学,以丰富的海量数据为基础,佐以实验的验证,有大量可供挖掘探索的科研课题,请师弟师妹们抓住机会大展宏图吧!
03 硕士论文简介
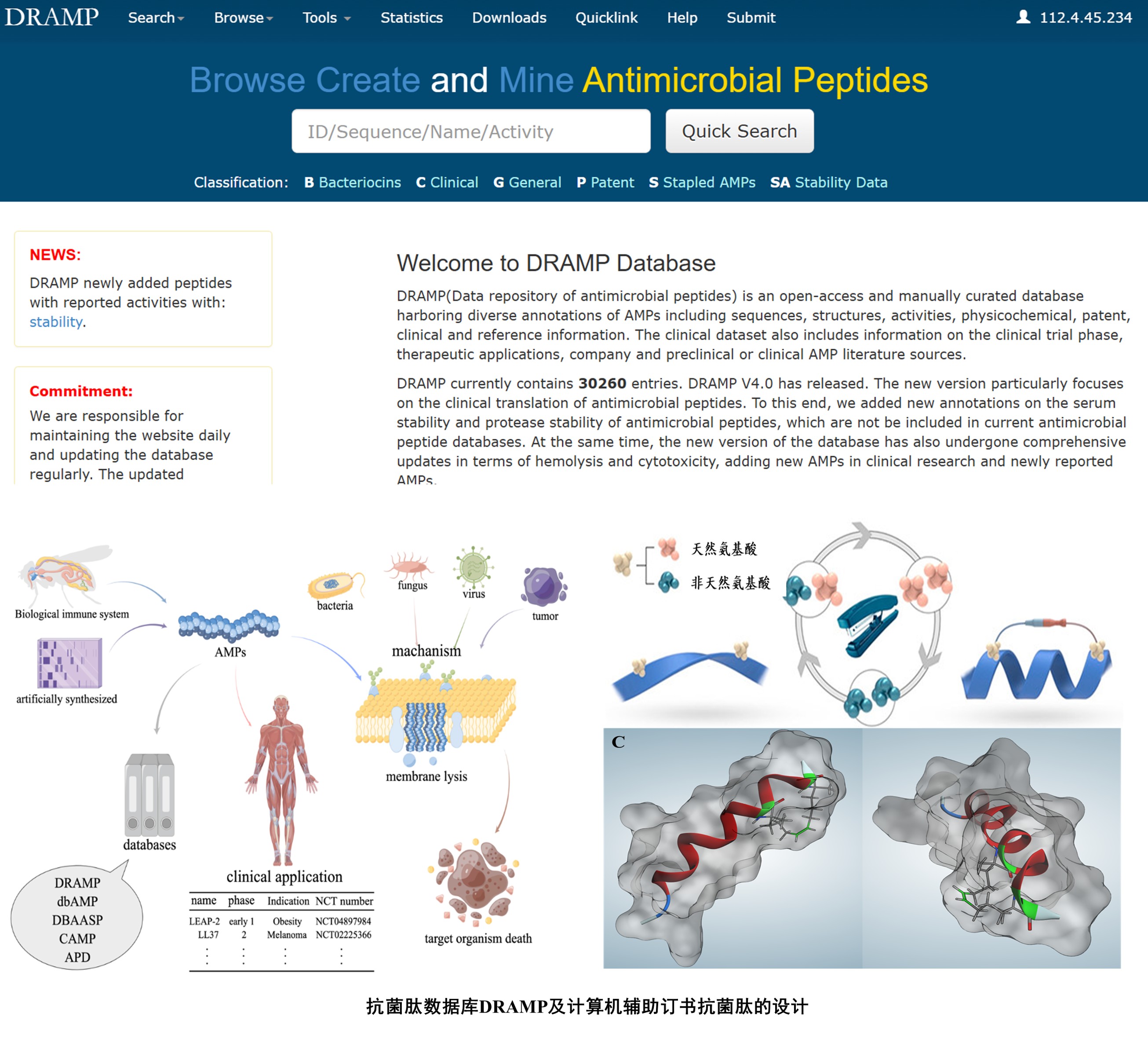
该论文主要涉及抗菌肽数据库更新和订书肽活性预测模型构建两大部分。由于抗生素耐药性问题日益严重,亟需新的抗菌药物,抗菌肽因其独特的作用机制和不易诱导耐药性被广泛关注。为促进新型抗菌肽设计,本论文主要完成的工作包括:首先,对DRAMP抗菌肽数据库进行了更新和维护,新增了2360条抗菌肽条目,其中包括181条订书肽。创新性地提出使用封闭式字母来标记修饰位点,使订书肽的环化位点可以直观展示。其次,利用AlphaFold2和RoseTTAFold预测了948个订书肽和含二硫键环化多肽的结构,并从中提取分子描述符特征。最后,构建了针对革兰氏阴性菌的抗菌活性预测模型,其中AutoGluon和GBDT模型表现最佳,准确率达到77.9%。基于这些模型,设计并筛选出5个潜在的Magainin II衍生订书抗菌肽。
本研究创新点主要在于:1.首次收集订书肽信息并构建其活性预测模型,为新型订书肽设计奠定了基础;2.率先提出使用封闭式字母表示订书氨基酸位点的新方法,为解决数据库记录修饰氨基酸残基难题提供了新的思路。这些工作为不仅可为新型抗菌肽的设计提供参考,还可为机器学习和人工智能在多肽药物设计中的应用提供帮助。
学术成果
课题组成功开发并更新了抗菌肽数据库DRAMP的3.0版本,相关论文发表在数据库领域的顶尖期刊Nucleic Acids Research上。作为一个手动注释的开放获取数据库,DRAMP 3.0新增了2360条记录,使总条目达到了22259条。这些条目包括5891条普通条目、16110条专利条目、77条临床条目以及181条新型环化抗菌肽条目。首次系统性地收集和组织了环化抗菌肽(stapled AMPs)的信息。这类肽通过化学修饰形成环状结构,具有良好的抗菌活性和结构稳定性。为了帮助设计低毒性的抗菌肽,我们还新增了细胞毒性数据。此外,数据库还包含263个预测结构和300多个链接到蛋白质数据库(PDB)的实验结构。对于数据库的更新还包括提供了新的分类系统,增加了细胞毒性、化学修饰、链间键、手性等新的注释字段等。促使DRAMP成为国内外最重要的抗菌肽数据库之一,在全球生物学数据库中位列前5%。
论文另一个重要成果是基于数据库中订书肽数据和信息,基于机器学习方法构建了抗革兰氏阴性菌的活性预测模型,其中AutoGluon和GBDT模型表现最佳,准确率达到77.9%。基于这些模型,设计并筛选出5个潜在的Magainin II衍生订书抗菌肽,为新型订书肽的研发奠定了基础,并可促进机器学习及人工智能技术在多肽药物设计领域的应用。

